NeuralKG是一个支持多种知识图谱表示学习/知识图谱嵌入(Knowledge Graph Embedding, KGE)模型的Python工具包,其中实现了多种传统知识图谱嵌入、基于图神经网络的知识图谱嵌入以及基于规则的知识图谱嵌入方法。同时为初学者提供了详细的文档以及一个开放共享的知识图谱表示学习社区网站。
本文利用此开源工具包实现简易、方便地在生物分子领域数据集上训练,仅需提供模型要求格式的数据集,设置所需模型、超参数即可开始训练。
安装方式详见NeuralKG。
数据介绍及处理
NeuralKG需要将数据处理为如下五个文件:
entities.dict
relations.dict
train.txt
valid.txt
test.txt
其中entities.dict和relations.dict每行为ID\t名称,ID为从0开始的序号;train.txt、valid.txt、test.txt每行为头节点\t关系\t尾节点
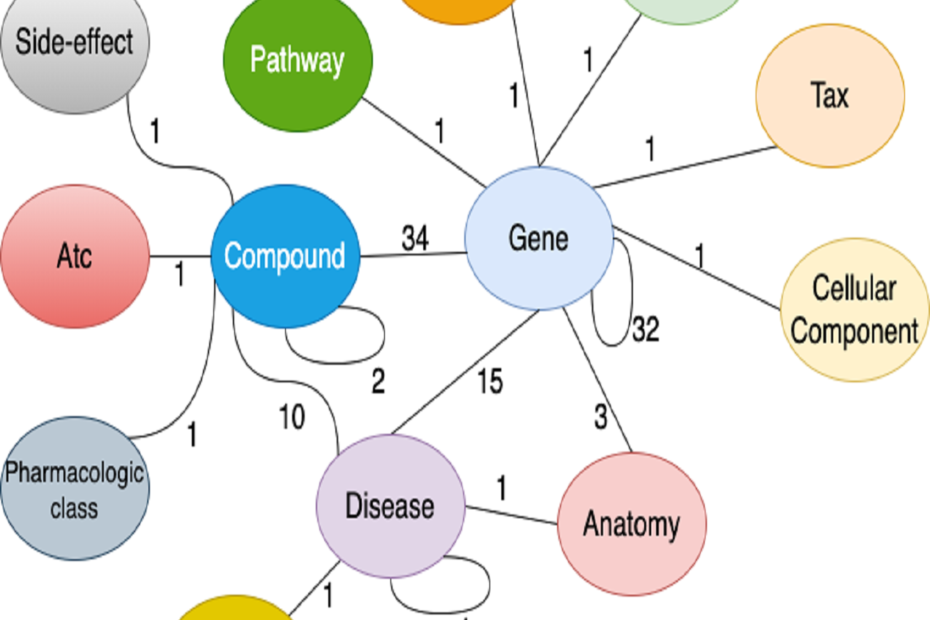
Drug Repurposing Knowledge Graph
大规模药物重定位知识图谱(DRKG)是一种涉及基因、化合物、疾病、生物学过程、副作用和症状的综合性生物知识图。它收集来自DrugBank、Hetionet、GNBR、String、IntAct 、DGIdb六个数据库的信息,以及从最近的出版物收集的数据,特别是与COVID19相关的数据。共包含13种实体类型的97238个实体,以及分属于107种关系类型的5874261个三元组数据。
他们还提供了使用GNN预训练DrugBank中的大多数小分子药物的嵌入向量。该预训练嵌入向量基于监督分子性质预测、与自监督学习方法相结合等不同方法训练获得,均可在其首页下载。
数据集的详细情况见GitHub,整理为三元组的数据集见下载链接,将其转换为所需格式即可。
配置文件
NeuralKG提供了两种配置参数的方式:使用YAML格式的配置文件调整各个参数,或者训练时用命令行调整。具体参数功能可见参数描述,可以很简单地通过litmodel_name选择传统KGE、基于GNN、基于Rule的相关KGE模型,并由model_name选择具体模型。
YAML配置文件可以参考GitHub中config的示例编写,运行方式:
python --load_config --config_path <your-config.yaml>
命令行可以参考GitHub中script的示例编写,运行方式:
bash <your-script.sh>
这里我们选择使用KGE模型中的TransE、ComplEx和Rotate进行相关实验测试模型。
实验结果
| Model | MRR | Hit@1 | Hit@3 | Hit@10 |
|---|---|---|---|---|
| TransE | 0.235 | 0.087 | 0.334 | 0.461 |
| ComplEx | 0.315 | 0.169 | 0.416 | 0.539 |
| RotatE | 0.278 | 0.142 | 0.370 | 0.486 |